Capítulo 35 Redes
35.1 Análise de redes
35.1.1 O que é análise de rede?
A análise de redes é uma abordagem estatística gráfica baseada na teoria dos grafos que permite representar, explorar e interpretar relações complexas entre múltiplas variáveis analisadas simultaneamente.320
Nessa abordagem, as variáveis são representadas por nodos (ou nós) e as relações entre elas por arestas, formando uma estrutura relacional que evidencia padrões de associação, interdependência e organização do sistema estudado.320
Diferentemente de métodos tradicionais, como análises univariadas ou modelos de regressão clássicos, a análise de redes não foca relações isoladas entre variáveis, mas sim o comportamento conjunto do sistema, permitindo observar fenômenos emergentes que não seriam detectáveis individualmente.320
A análise de redes representa uma mudança conceitual importante em relação às abordagens estatísticas tradicionais, ao enfatizar sistemas, interações e complexidade.320
35.1.2 Por que a análise de redes é útil em pesquisa científica?
Muitos fenômenos científicos, especialmente nas ciências da saúde, são multifatoriais, interdependentes e não lineares, envolvendo variáveis biológicas, comportamentais, psicológicas e sociais.320
Análises univariadas tendem a simplificar esses fenômenos ao avaliar efeitos médios ou relações diretas entre pares de variáveis, o que pode ocultar padrões relevantes.320
A análise de redes permite: visualizar associações simultâneas entre diversas variáveis; identificar variáveis centrais em um sistema; detectar subestruturas densas ou agrupamentos de variáveis fortemente associadas; explorar potenciais mecanismos intermediários ou mediadores.320
35.1.3 Quais são as limitações da análise de redes?
Apesar de seu potencial, a interpretação dos resultados deve ser cuidadosa, especialmente em estudos observacionais, evitando inferências causais indevidas.320
35.2 Matriz de incidência
35.2.1 O que é uma matriz de incidência?
Uma matriz de incidência é uma representação tabular que descreve a relação entre dois conjuntos distintos de entidades, como nodos e arestas em uma rede.320
Em uma matriz de incidência, as linhas representam um conjunto de entidades (nodos) e as colunas representam outro conjunto (arestas). A presença ou ausência de uma relação entre as entidades é indicada por valores binários (0 ou 1) ou por pesos numéricos que refletem a intensidade da relação.320
| Sexo | Tabagismo | Atividade |
|---|---|---|
| F | Sim | Baixa |
| M | Não | Alta |
| F | Não | Moderada |
| F | Sim | Baixa |
| M | Não | Alta |
Code
# Exemplo dados categóricos
dados_cat <- data.frame(
Sexo = c("F", "M", "F", "F", "M"),
Tabagismo = c("Sim", "Não", "Não", "Sim", "Não"),
Atividade = c("Baixa", "Alta", "Moderada", "Baixa", "Alta")
)
# Matriz de incidência (one-hot encoding)
matriz_incidencia <- model.matrix(
~ Sexo + Tabagismo + Atividade - 1,
data = dados_cat
)
# Converter para data.frame
matriz_incidencia <- as.data.frame(matriz_incidencia)
# exibe a tabela de dados
knitr::kable(
matriz_incidencia,
align = "c",
format = ifelse(knitr::is_html_output(), "html", "latex"),
booktabs = TRUE,
linesep = "",
escape = FALSE
) %>%
kableExtra::kable_styling(
latex_options = c("basic"),
bootstrap_options = c("basic", "hover", "condensed", "responsive"),
full_width = ifelse(knitr::is_html_output(), T, T),
position = "center"
) %>%
kableExtra::row_spec(0, bold = TRUE, extra_css = "border-top: 1px solid; border-bottom: 1px solid") %>%
kableExtra::column_spec(1, bold = TRUE) %>%
kableExtra::row_spec(dim(matriz_incidencia)[1], extra_css = "border-bottom: 1px solid")| SexoF | SexoM | TabagismoSim | AtividadeBaixa | AtividadeModerada |
|---|---|---|---|---|
| 1 | 0 | 1 | 1 | 0 |
| 0 | 1 | 0 | 0 | 0 |
| 1 | 0 | 0 | 0 | 1 |
| 1 | 0 | 1 | 1 | 0 |
| 0 | 1 | 0 | 0 | 0 |
O pacote stats134 fornece a função model.matrix para criar uma matrix de incidência por expansão de variáveis indicadoras.
35.3 Elementos da rede
35.3.1 Quais são os principais elementos de uma rede?
Nodos (nós): representam as variáveis do estudo, como sintomas, doenças, características clínicas, sociais ou psicológicas.320
Arestas: representam as relações entre os nodos. Podem indicar correlação, associação parcial, dependência condicional ou outro tipo de relação estatística.320
Peso das arestas: em redes ponderadas, a espessura da aresta indica a magnitude da relação; relações mais fortes são representadas por conexões mais espessas.320
Sinal das arestas: geralmente codificado por cores, indicando associações positivas ou negativas.320
Code
# Definir arestas com peso e sinal
edges <- data.frame(
from = c("A", "A", "B", "C", "D"),
to = c("B", "C", "C", "D", "A"),
peso = c( 0.8, -0.5, 0.6, -0.7, 0.4)
)
g <- igraph::graph_from_data_frame(edges, directed = FALSE)
# Peso absoluto define espessura da aresta
igraph::E(g)$width <- abs(igraph::E(g)$peso) * 4
# Cor indica sinal da relação
igraph::E(g)$color <- ifelse(igraph::E(g)$peso > 0, "steelblue", "firebrick")
# Rótulo das arestas (valor do peso)
igraph::E(g)$label <- round(igraph::E(g)$peso, 2)
igraph::E(g)$label.color <- "black"
igraph::E(g)$label.cex <- 0.8
# Nós
igraph::V(g)$size <- 30
igraph::V(g)$color <- "grey85"
igraph::V(g)$frame.color <- "black"
igraph::V(g)$label.cex <- 1.1
igraph::V(g)$label.color <- "black"
set.seed(123)
layout_fr <- igraph::layout_with_fr(g)
plot(
g,
layout = layout_fr,
vertex.label = igraph::V(g)$name,
edge.label = igraph::E(g)$label
)
legend(
"topleft",
inset = 0.02,
title = "Legend",
legend = c(
"Node (variable)",
"Positive edge",
"Negative edge",
"Edge width ∝ |weight|"
),
pch = c(21, NA, NA, NA),
pt.bg = c("grey85", NA, NA, NA),
lty = c(NA, 1, 1, 1),
lwd = c(NA, 2, 2, 4),
col = c("black", "steelblue", "firebrick", "black"),
bty = "n",
cex = 0.9
)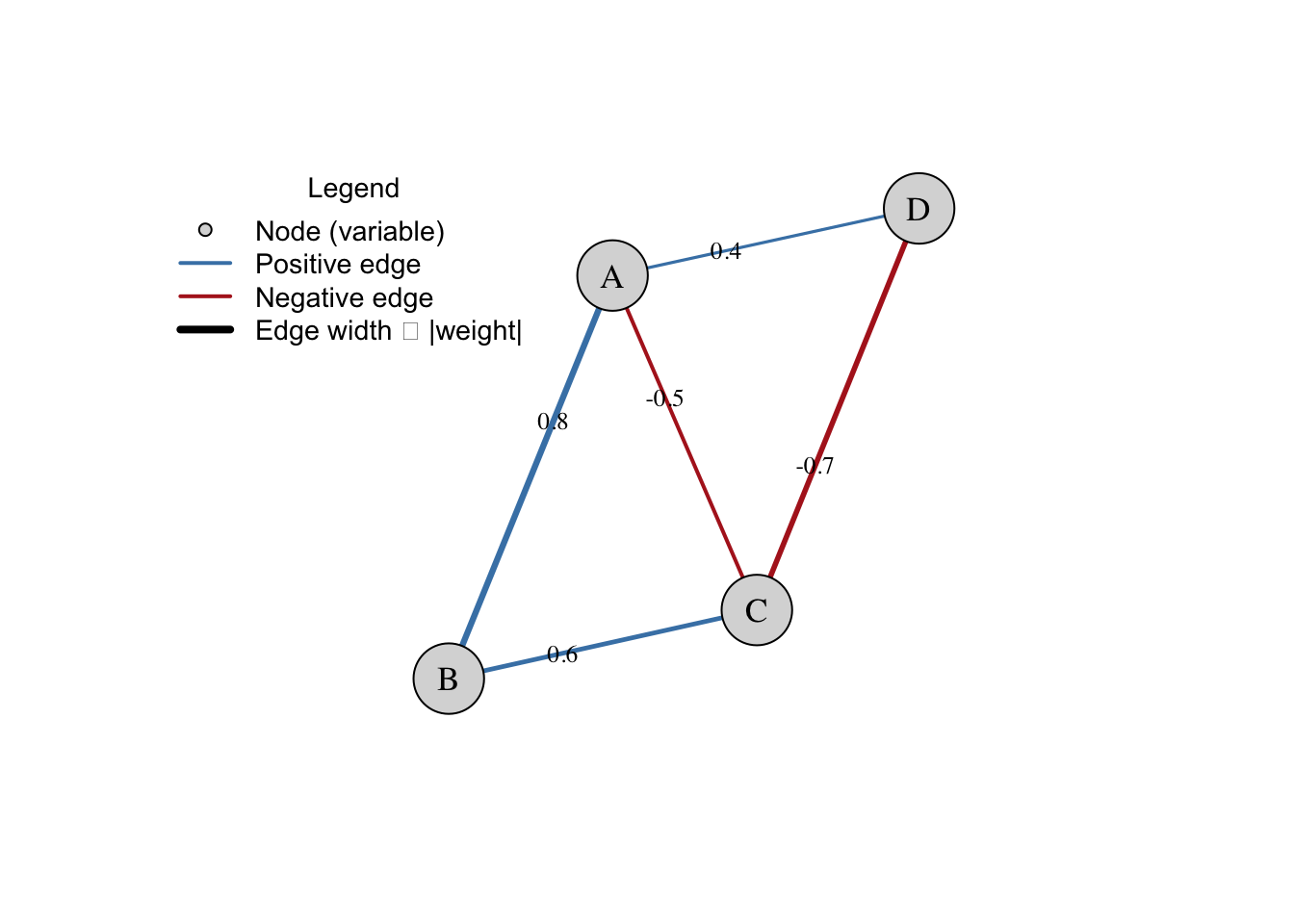
Figura 35.1: Exemplo de grafo de rede com nodos e arestas ponderadas.
35.3.2 Como as redes podem ser classificadas?
Redes não ponderadas: indicam apenas a presença ou ausência de relação entre os nodos.320
Redes ponderadas: representam também a intensidade da relação.320
Redes direcionais: possuem setas nas arestas, indicando direção da relação (por exemplo, causalidade hipotética).320
Redes não direcionais: não assumem direção causal e são mais apropriadas para estudos observacionais e transversais.320
Em pesquisas transversais, especialmente em saúde, redes não direcionais e ponderadas são geralmente preferidas, pois evitam inferências causais indevidas quando não há informação temporal adequada.320
35.3.3 O que define a posição dos nodos em um grafo de rede?
A posição espacial dos nodos é determinada por algoritmos de disposição (layout), que organizam a rede de modo a facilitar a interpretação visual.320
Um dos algoritmos mais utilizados é o Fruchterman–Reingold321, que simula forças de atração e repulsão entre os nodos, posicionando nodos mais fortemente associados mais próximos entre si. A proximidade entre nodos em um grafo geralmente reflete maior associação estatística.320
35.4 Tipos de redes
35.4.1 Quais são os principais tipos de redes estatísticas?
Redes de correlação: baseadas em matrizes de correlação simples; são fáceis de interpretar, mas podem conter associações espúrias.320
Redes de correlação parcial: representam relações entre dois nodos controlando todas as demais variáveis do sistema, reduzindo associações indiretas.320
Modelos gráficos gaussianos: uma forma específica de rede de correlação parcial, muito utilizada em psicometria e epidemiologia.320
Modelos gráficos mistos: permitem a análise conjunta de variáveis contínuas, ordinais e dicotômicas, comuns em dados reais de saúde.320
35.4.2 Como reduzir associações espúrias em redes?
Em conjuntos de dados com muitas variáveis, redes podem se tornar densas e difíceis de interpretar. Para contornar esse problema, utiliza-se penalização estatística, como o método LASSO (Least Absolute Shrinkage and Selection Operator).320
Na análise de redes, o Graphical LASSO (gLASSO) elimina associações fracas, resultando em redes mais parcimoniosas e interpretáveis.320
35.5 Métricas de rede
35.5.1 O que são medidas de centralidade?
Medidas de centralidade quantificam a importância relativa de cada nodo no sistema.320
Grau: número de conexões diretas de um nodo.320
Força: soma dos pesos das conexões de um nodo, sendo uma das medidas mais utilizadas em redes ponderadas.320
Intermediação: frequência com que um nodo atua como ponte entre outros nodos.320
Proximidade: quão próximo um nodo está de todos os outros.320
Agrupamento: tendência de um nodo formar grupos com seus vizinhos.320
Code
edges <- data.frame(
from = c("A", "A", "B", "C", "D"),
to = c("B", "C", "C", "D", "A"),
peso = c( 0.8, -0.5, 0.6, -0.7, 0.4)
)
g <- igraph::graph_from_data_frame(edges, directed = FALSE)
# Separar semântica
igraph::E(g)$peso <- edges$peso
igraph::E(g)$abs_peso <- abs(edges$peso)
# Força dos nós
forca <- igraph::strength(g, weights = igraph::E(g)$abs_peso)
# Escalar tamanho do nó pela força
igraph::V(g)$size <- 20 + 30 * (forca / max(forca))
# Cor neutra para nós
igraph::V(g)$color <- "grey85"
igraph::V(g)$frame.color <- "black"
# Arestas
igraph::E(g)$width <- igraph::E(g)$abs_peso * 4
igraph::E(g)$color <- ifelse(igraph::E(g)$peso > 0, "steelblue", "firebrick")
# Layout correto (somente pesos positivos)
set.seed(123)
layout_fr <- igraph::layout_with_fr(g, weights = igraph::E(g)$abs_peso)
plot(
g,
layout = layout_fr,
vertex.label = paste0(igraph::V(g)$name),
edge.label = round(igraph::E(g)$peso, 2)
)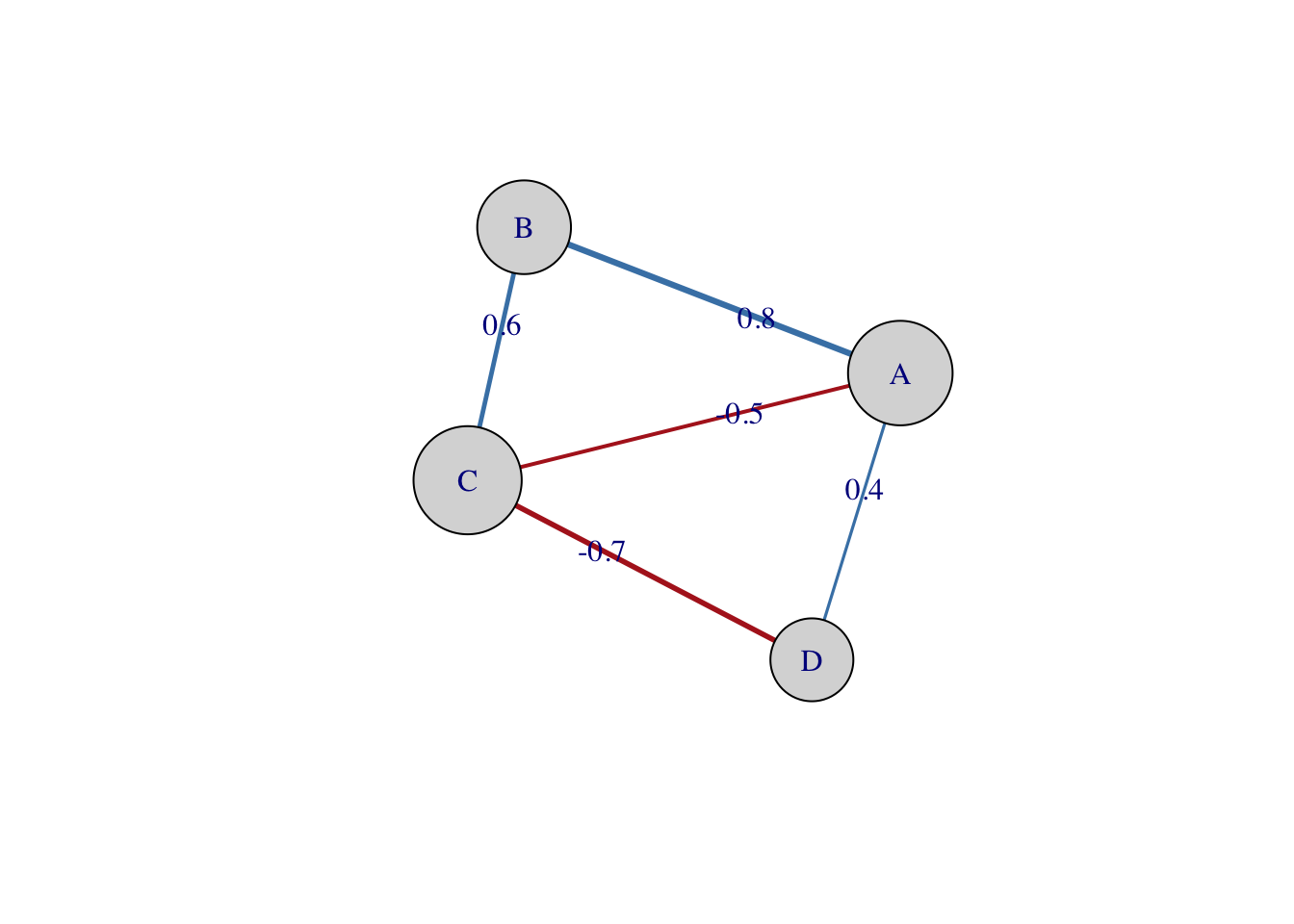
Figura 35.2: Exemplo de grafo de rede com tamanho dos nodos proporcional à força.
Code
edges <- data.frame(
from = c("A", "A", "B", "C", "D"),
to = c("B", "C", "C", "D", "A"),
peso = c( 0.8, -0.5, 0.6, -0.7, 0.4)
)
g <- igraph::graph_from_data_frame(edges, directed = FALSE)
grau <- igraph::degree(g)
forca <- igraph::strength(g, weights = igraph::E(g)$abs_peso)
intermediacao <- igraph::betweenness(
g,
weights = 1 / igraph::E(g)$abs_peso,
normalized = TRUE
)
proximidade <- igraph::closeness(
g,
weights = 1 / igraph::E(g)$abs_peso,
normalized = TRUE
)
agrupamento <- igraph::transitivity(
g,
type = "local",
isolates = "zero"
)
# Converter para data.frame
metricas <- data.frame(
Nó = names(grau),
Grau = grau,
Força = round(forca, 2),
Intermediação = round(intermediacao, 3),
Proximidade = round(proximidade, 3),
Agrupamento = round(agrupamento, 3)
)
# exibe a tabela de dados
knitr::kable(
metricas,
align = "c",
format = ifelse(knitr::is_html_output(), "html", "latex"),
booktabs = TRUE,
linesep = "",
escape = FALSE
) %>%
kableExtra::kable_styling(
latex_options = c("basic"),
bootstrap_options = c("basic", "hover", "condensed", "responsive"),
full_width = ifelse(knitr::is_html_output(), T, T),
position = "center"
) %>%
kableExtra::row_spec(0, bold = TRUE, extra_css = "border-top: 1px solid; border-bottom: 1px solid") %>%
kableExtra::column_spec(1, bold = TRUE) %>%
kableExtra::row_spec(dim(metricas)[1], extra_css = "border-bottom: 1px solid")| Nó | Grau | Força | Intermediação | Proximidade | Agrupamento | |
|---|---|---|---|---|---|---|
| A | A | 3 | 3 | 0.167 | 1.00 | 0.667 |
| B | B | 2 | 2 | 0.000 | 0.75 | 1.000 |
| C | C | 3 | 3 | 0.167 | 1.00 | 0.667 |
| D | D | 2 | 2 | 0.000 | 0.75 | 1.000 |
O pacote igraph322 fornece a função graph_from_incidence_matrix para criar uma rede a partir de uma matriz de incidência.
O pacote bootnet323 fornece a função bootnet para avaliação da estabilidade e precisão das redes por reamostragem.
Ferreira, Arthur de Sá. Ciência com R: Perguntas e respostas para pesquisadores e analistas de dados. Rio de Janeiro: 1a edição,