Capítulo 4 Pensamento computacional
4.1 R
4.1.1 O que é R?
R é um programa de computador de código aberto com linguagem computacional direcionada para análise estatística.32,33
R version 4.5.2 (2025-10-31) está disponível gratuitamente em Comprehensive R Archive Network (CRAN).34
4.1.2 Por que usar R?
- R é o software de maior abrangência de métodos estatísticos, possui sintaxe que permite análises estatísticas reproduzíveis e está disponível gratuitamente no Comprehensive R Archive Network (CRAN).28,34
4.1.3 O que é R Markdown?
R Markdown35 é uma ferramenta que permite a integração de texto, código e saída em um único documento.REF?
O R Markdown é uma extensão do Markdown, que é uma linguagem de marcação simples e fácil de aprender, que é usada para formatar texto.REF?
O R Markdown permite a inclusão de blocos de código R, Python, SQL, C++, entre outros, e a saída desses blocos de código é incorporada ao documento final.REF?
O R Markdown é uma ferramenta poderosa para a criação de relatórios dinâmicos, que podem ser facilmente atualizados com novos dados ou análises.REF?
O R Markdown é amplamente utilizado na comunidade científica para a criação de relatórios de pesquisa, artigos científicos, apresentações, livros, entre outros.REF?
O trabalho com RMarkdown35 permite um fluxo de dados totalmente transparente, desde o conjunto de dados coletados até o manuscrito finalizado. Todos os aspectos do fluxo de dados podem ser incorporados em blocos de R script (chunk), exibindo tanto o R script quando o respectivo texto, tabelas e figuras formatadas no estilo científico de interesse.36
O RMarkdown35 foi projetado especificamente para relatórios dinâmicos onde a análise é realizada em R e oferece uma flexibilidade incrível por meio de uma linguagem de marcação.28
4.2 RStudio
4.2.1 O que é RStudio?
RStudio é um ambiente de desenvolvimento integrado (integrated development environment, IDE) desenvolvido visando a reprodutibilidade e a simplicidade para a criação e disseminação de conhecimento.33,41
O ambiente do RStudio é dividido em paineis:
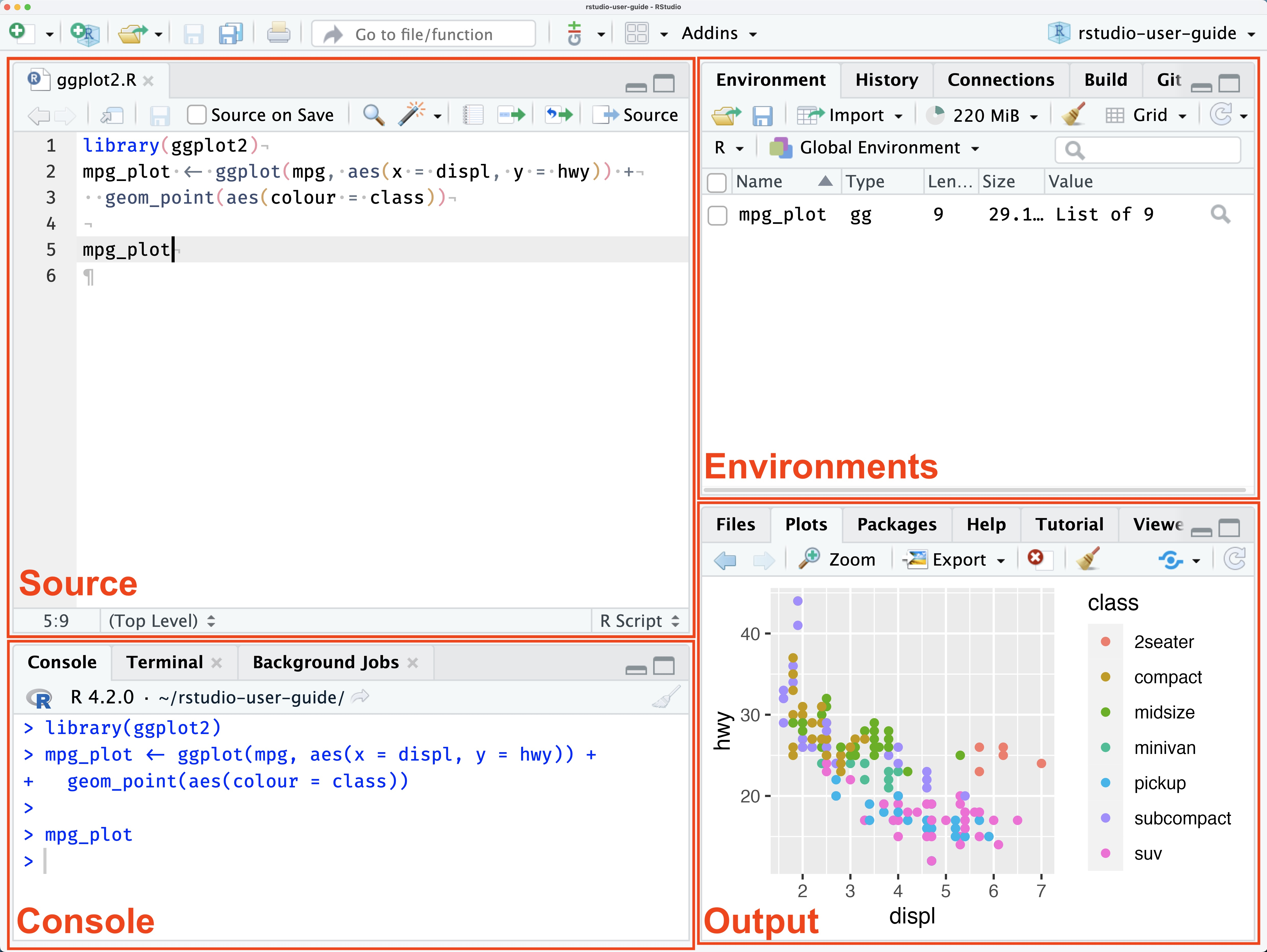
Figura 4.1: Interface do RStudio. Fonte: https://docs.posit.co/ide/user/
As principais características do RStudio incluem um ambiente de edição com abas para acesso rápido a arquivos, comandos e resultados; histórico de comandos previamente utilizados; ferramentas para visualização de bancos de dados e elaboração de scripts e gráficos e tabelas.33,41
RStudio está disponível gratuitamente em Posit.
O pacote learnr42 fornece tutoriais interativos para RStudio.
4.3 Scripts
4.3.1 O que são R scripts?
“Scripts são dados”.29
Scripts permitem ao usuário se concentrar nas tarefas mais importantes da computação e utilizar pacotes ou bibliotecas para executar as funções mais básicas com maior eficiência.29
Um script é um arquivo de texto contendo (quase) os mesmos comandos que você digitaria na linha de comando do R. O “quase” refere-se ao fato de que se você estiver usando sink() para enviar a saída para um arquivo, você terá que incluir alguns comandos em print() para obter a mesma saída da linha de comando.REF?
Code
# Exemplo de R script
# Este é um comentário
# Esta é uma variável
variavel <- 3.14 # Atribui o valor 3.14 à variável
# Esta é uma função
f <- function(x) {
return(x^2) # Retorna o quadrado do valor de x
}
# Esta é uma chamada de função
resultado <- f(variavel) # Chama a função f com a variável como argumento
# Exibe o resultado da função
print(resultado) # Exibe o resultado na saída padrão
# Este é um vetor
vetor <- c(1, 2, 3, 4, 5) # Cria um vetor com os valores de 1 a 5
# Exibe o vetor
print(vetor) # Exibe o vetor na saída padrão
# Esta é uma matrix
matriz <- matrix(1:9, nrow=3, ncol=3) # Cria uma matriz 3x3 com os valores de 1 a 9
# Exibe a matriz
print(matriz) # Exibe a matriz na saída padrão
# Esta é uma lista
lista <- list(nome="João", idade=30, altura=1.75) # Cria uma lista com nome, idade e altura
# Exibe a lista
print(lista) # Exibe a lista na saída padrão
# Este é um dataframe
dataframe <- data.frame(nome=c("João", "Maria", "José"), idade=c(30, 25, 40), altura=c(1.75, 1.60, 1.80)) # Cria um dataframe com nome, idade e altura
# Exibe o dataframe
print(dataframe) # Exibe o dataframe na saída padrão
# Este é um loop for
for (i in 1:5) {
print(i) # Exibe os valores de 1 a 5 na saída padrão
}
# Este é um loop while
j <- 1
while (j <= 5) {
print(j) # Exibe os valores de 1 a 5 na saída padrão
j <- j + 1 # Incrementa o valor de j em 1
}
# Este é um condicional if-else
k <- 3
if (k > 0) {
print("k é positivo") # Exibe "k é positivo" se k for maior que 0
} else if (k < 0) {
print("k é negativo") # Exibe "k é negativo" se k for menor que 0
} else {
print("k é zero") # Exibe "k é zero" se k for igual a 0
}
# Fim do exemplo de R script4.3.2 Quais são as boas práticas na redação de scripts?
Use nomes consistentes para as variáveis.43
Defina os tipos de variáveis adequadamente no banco de dados.43
Defina constantes — isto é, variáveis de valor fixo — ao invés de digitar valores.43
Use e cite os pacotes disponíveis para suas análises.43
Teste o script antes de sua utilização.43
Conduza revisão por pares do código durante a redação (digitação em dupla).43
O pacote formatR45 fornece a função tidy_source para formatar um R script.
O pacote styler46 fornece a função style_file para formatar um R script.
O pacote lintr47 fornece a função lint para verificar a adesão de um script a um determinado estilo, identificando erros de sintaxe e possíveis problemas semânticos.
4.4 Pacotes
4.4.1 O que são pacotes?
Pacotes são conjuntos de scripts programados pela comunidade e compartilhados para uso público.33
Os pacotes ficam armazenados no Comprehensive R Archive Network (CRAN) e podem ser instalados diretamente no RStudio.33,34
Na mais recente atualização deste livro, o [Comprehensive R Archive Network (CRAN) possui 392513 pacotes disponíveis.33,34
Os pacotes disponíveis podem ser encontrados em R PACKAGES DOCUMENTATION.48
O pacote utils49 fornece a função install.packages para instalar os pacotes no computador.
O pacote utils49 fornece a função installed.packages para listar os pacotes instalados no computador.
O pacote utils49 fornece a função update.packages para atualizar os pacotes instalados no computador.
O pacote roxygen250 fornece a função roxygenize para criar arquivos .Rd para documentar pacotes.
4.5 Aplicativos
4.5.1 O que são Shiny Apps?
- Shiny Apps são aplicativos web interativos que permitem a criação de interfaces gráficas para visualização e análise de dados em tempo real, utilizando o R como backend.REF?
4.6 Manuscritos reproduzíveis
4.6.1 O que são manuscritos reproduzíveis?
- Manuscritos reproduzíveis — manuscritos executáveis ou relatórios dinâmicos — permitem a produção de um manuscrito completo a partir da integração do banco de dados da(s) amostra(s), do(s) script(s) de análise estatística (incluindo comentários para sua interpretação), dos pacotes ou bibliotecas utilizados, das fontes e referências bibliográficas citadas, além dos demais elementos textuais (tabelas, gráficos) - todos gerados dinamicamente.29
4.6.2 Por que usar manuscritos reproduzíveis?
No processo tradicional de redação científica há muitas etapas de copiar e colar não reproduzíveis envolvidas. Documentos dinâmicos combinam uma ferramenta de processamento de texto com o R script que produz o texto/tabela/figura a ser incorporado no manuscrito.28
Ao trabalhar com relatórios dinâmicos, é possível extrair o mesmo script usado para análise estatística. Os documentos podem ser compilados em vários formatos de saída e salvos como DOCX, PPTX e PDF.28
Muitos erros de análise poderiam ser evitados com a adoção de boas práticas de programação em manuscritos reproduzíveis.51
O pacote rmarkdown35 fornece as funções render para criar manuscritos reprodutíveis a partir de arquivos .Rmd.
O pacote officedown52 fornece as funções rdocx_document e rpptx_document para criar arquivos DOCX e PPTX, respectivamente, com o conteúdo criado no manuscrito reprodutível.
O pacote bookdown53 fornece as funções gitbook, pdf_book, epub_book e html_document2 para criar documentos reprodutíveis em diversos formatos (Git, PDF, EPUB e HTML, respectivamente).
4.6.3 Como manuscritos reprodutíveis contribuem para a ciência?
- O compartilhamento de bancos de dados e seus scripts de análise estatística permitem a adoção de práticas reprodutíveis, tais como a reanálise dos dados.54
O pacote projects55 fornece a função setup_projects para criar um projeto com arquivos organizados em diretórios.
O pacote rmarkdown35 fornece a função render para criar manuscritos reprodutíveis a partir de arquivos .Rmd.
O pacote bookdown53 fornece as funções gitbook, pdf_book, epub_book e html_document2 para criar documentos reprodutíveis em diversos formatos (Git, PDF, EPUB e HTML, respectivamente).
4.7 Compartilhamento
4.7.1 Por que compartilhar scripts?
- Compartilhar o script — principalmente junto aos dados — pode facilitar a replicação direta do estudo, a detecção de eventuais erros de análise, a detecção de pesquisas fraudulentas.56
4.7.2 O que pode ser compartilhado?
Idealmente, todos os scripts, pacotes/bibliotecas e dados necessários para outros reproduzirem seus dados.44
Minimamente, partes importantes incluindo implementações de novos algoritmos e dados que permitam reproduzir um resultado importante.44
4.7.4 Como preparar scripts para compartilhamento?
Providencie a documentação sobre seu script (ex.: arquivo README).44
Inclua a versão dos pacotes usados no seu script por meio de um script inicial para instalação de pacotes (ex.: ‘instalar.R’).51
Documente em um arquivo README os arquivos disponíveis e os pré-requisitos necessários para executar o código (ex.: pacotes e respectivas versões). Uma lista de configurações (hardware e software) que foram usadas para rodar o código pode ajudar na reprodução dos resultados.27
Use endereços de arquivos relativos.51
Crie links persistentes para versões do seu script.44
Defina uma semente para o gerador de números aleatórios em scripts com métodos computacionais que dependem da geração de números pseudoaleatórios.27
O pacote base57 fornece a função set.seed para especificar uma semente para reprodutibilidade de computações que envolvem números aleatórios.
Escolha uma licença apropriada para garantir os direitos de criação e como outros poderão usar seus scripts.44
Teste o script em uma nova sessão antes de compartilhar.51
Cite todos os pacotes relacionados à sua análise.58
O pacote grateful59 fornece a função cite_packages para citar os pacotes utilizados em um projeto R.
- Inclua a informação da sessão em que os scripts foram rodados.51
O pacote utils49 fornece a função sessionInfo para descrever as características do programa, pacotes e plataforma da sessão atual.
4.7.5 O que incluir no arquivo README?
Título do trabalho.27
Autores do trabalho.27
Principais responsáveis pela escrita do script e quaisquer outras pessoas que fizeram contribuições substanciais para o desenvolvimento do script.27
Endereço de e-mail do autor ou contribuidor a quem devem ser direcionadas dúvidas, comentários, sugestões e bugs sobre o script.27
Lista de configurações nas quais o script foi testado, tais com nome e versão do programa, pacotes e plataforma.27
Ferreira, Arthur de Sá. Ciência com R: Perguntas e respostas para pesquisadores e analistas de dados. Rio de Janeiro: 1a edição,